研究论文
宏基因组分析揭示进展期腺瘤患者与结直肠癌患者肠道微生物组的不同特征
这项研究通过对26例晚期腺瘤(AA)患者和26例结直肠癌(CRC)患者的粪便样本进行宏基因组测序分析,旨在寻找能区分二者的微生物靶点,探索基于粪便微生物识别CRC的最佳方法。
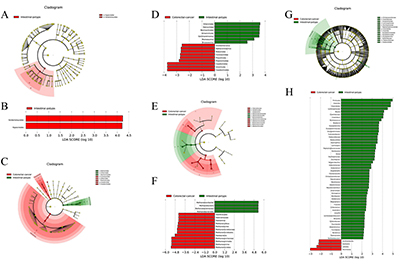
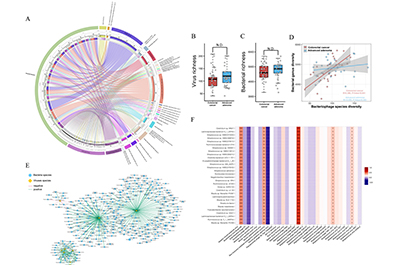
微生物群落差异:研究发现,AA和CRC患者肠道微生物群落的结构和多样性存在差异。在细菌、真菌、古菌和病毒的属水平上,多种微生物的丰度在两组间表现出不同,如CRC患者中志贺氏菌、曲霉、甲烷短杆菌等丰度有变化。LEfSe分析还筛选出了多个差异微生物标志物,且微生物与KEGG通路的相关性在两组间也有所不同。
预测模型评估:通过构建多种预测模型发现,基于15种关键病毒的随机森林(RF)模型识别CRC和AA的准确率为86.54%,而基于微生物单核苷酸多态性(SNP)的RF模型准确率高达92.31%,SNP受环境因素影响小。这表明微生物SNP在识别CRC方面表现更优,为区分AA和CRC提供了新的潜在方向,但研究存在样本量小等局限。