研究论文
使用全基因组测序绘制不同地理位置的结直肠癌相关肠道微生物谱
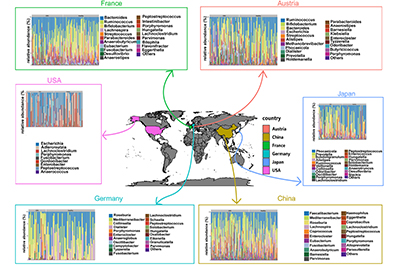
这项研究收集了来自6个国家的601个样本的宏基因组数据集,运用多种分析方法,旨在明确不同地区结直肠癌(CRC)患者的肠道微生物组成和结构差异,并构建基于区域差异的CRC风险预测模型。
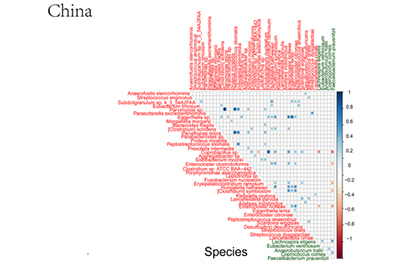
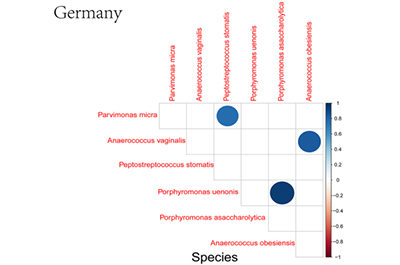
不同区域肠道菌群差异:研究发现,不同区域的肠道细菌群落组成存在差异,不同国家CRC患者的差异肠道细菌也不一致,但在物种水平上,所有国家都存在一些共同的细菌,如口腔消化链球菌和具核梭杆菌。此外,口腔消化链球菌在不同区域都与其他细菌存在相关性,且在属水平上,消化链球菌也与其他细菌有关联 。
风险预测模型分析:构建的CRC风险预测模型显示,区域对模型准确性影响较小,基于所有区域样本构建的模型中,口腔消化链球菌和消化链球菌分别是物种和属水平上的重要变量。单一区域模型应用于其他区域时,准确性虽有波动但仍在可接受范围内,表明肠道微生物用于预测CRC风险不受区域差异的显著影响。