研究论文
基于肠道细菌的结直肠癌淋巴结转移预测模型
这项研究通过对119例有淋巴结转移和110例无淋巴结转移的结直肠癌患者的粪便样本进行肠道细菌16SrRNA检测和生物信息学分析,并构建预测模型,旨在筛选结直肠癌淋巴结转移的差异肠道细菌。
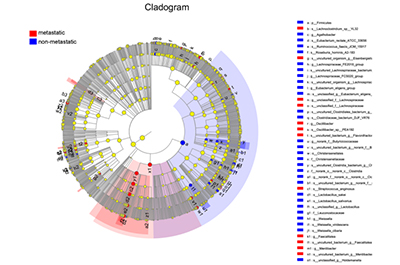
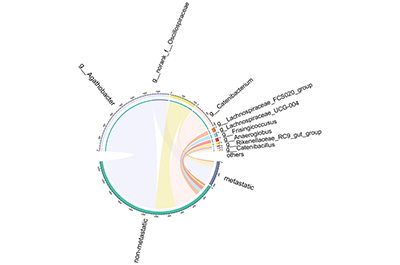
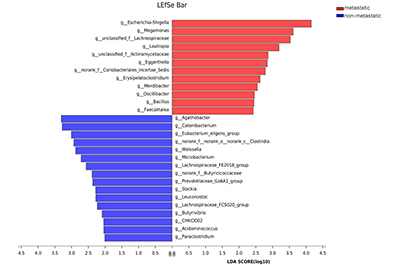
菌群差异及相关性:研究发现,两组患者肠道细菌群落丰富度和属水平的群落多样性基本一致。在属水平上筛选出 12 种差异细菌,如阿氏菌属、链形杆菌属等,这些细菌在无淋巴结转移的患者中相对丰度更高,且它们与无淋巴结转移的关联更紧密,不过两组细菌在功能上无显著差异。
预测模型构建结果:构建的预测模型显示,在发现集中,随机森林(RF)模型预测准确性最高,AUC为1.00,正确率达98.89%;在测试集中,支持向量机(SVM)模型表现最佳,AUC为0.73,正确率为72.92%。其中,不同模型中起关键作用的变量有所不同,如Lachnospiraceae_FCS020_group 在 RF、GBDT和Catboost模型中很重要,而 Lachnospiraceae_UCG-004在SVM模型中最为关键。